INTRODUCCIÓN
CRISPR (en inglés clustered regularly interspaced short palindromic repeats, en español repeticiones palindrómicas cortas agrupadas y regularmente interespaciadas).
Desde 2013 el sistema CRISPR/Cas se ha utilizado para la edición de genes. CRISPR-Cas es una herramienta de edición del ADN capaz de introducir cambios en el genoma, tras reconocer de forma específica la secuencia que se quiere modificar. Dicho con otras palabras, CRISPR-Cas es una tecnología que actúa como unas tijeras moleculares capaces de cortar una secuencia de ADN del genoma de forma específica e introducir, posteriormente, alguna modificación de interés.
El sistema de edición genómica CRISPR-Cas12b es una estrategia alternativa más eficiente que otros CRISPR a utilizar en células humanas.
DESCRIPCIÓN DE LA NUEVA TÉCNICA
CRISPR-Cas12 para la detección de SARS-CoV-2 a partir de ARN de muestra extraida de pacientes, llamado Reportador trans CRISPR dirigido por endonucleasa de ADN SARS-CoV-2 (DETECTR).
El ensayo DETECTR basado en CRISPR ofrece una alternativa visual y más rápida al ensayo de RT-PCR en tiempo real SARS-CoV-2 de los Centros para el Control y la Prevención de Enfermedades de los EE. UU., con un acuerdo predictivo positivo del 95% y un acuerdo predictivo negativo del 100%.(sin referencia estándar)
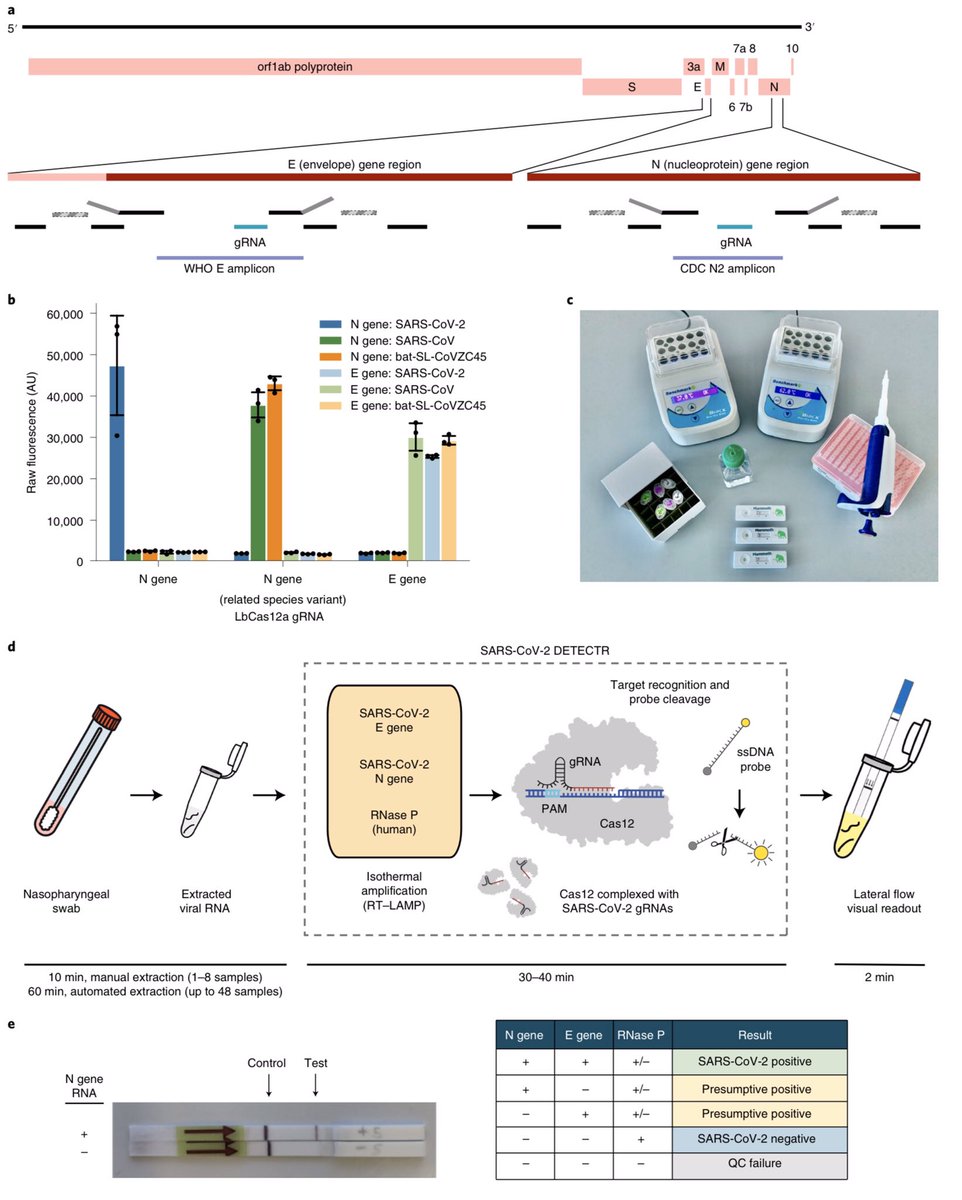
Mapa del genoma que muestra cebadores, sondas y ARNg. Visualización de cebadores y sondas en el genoma del SARS-CoV-2. Los cebadores RT-LAMP se indican mediante rectángulos negros, la posición de unión de la mitad F1c y B1c del cebador interno delantero (FIP) (gris) está representada por un rectángulo rayado con bordes punteados. b, especificidad de gRNA. Los gRNA Cas12 están programados para atacar específicamente el SARS-CoV-2 o detectar ampliamente las cepas de coronavirus relacionadas. El gen de ARN N utilizado en el ensayo (izquierda) fue específico para SARS-CoV-2, mientras que el gen de ARN E pudo detectar tres cepas de coronavirus similares al SARS (derecha). Un ARNg de gen N separado diseñado para atacar el SARS-CoV y un coronavirus murciélago no pudieron detectar el SARS-CoV-2 (centro). c, El equipo mínimo necesario para ejecutar el protocolo. Con los requisitos apropiados de nivel 2 de bioseguridad, el equipo mínimo requerido para ejecutar el protocolo después de la extracción de ARN incluye tubos Eppendorf con reactivos, bloques de calor o baño de agua (37 ° C y 62 ° C), agua libre de nucleasas, pipetas y puntas y flujo lateral tiras. d, Esquema del flujo de trabajo DETECTR del SARS-CoV-2. La extracción de ARN convencional se puede utilizar como entrada para DETECTR (preamplificación LAMP y detección basada en Cas12 para el gen E, el gen N y la RNasa P), que se visualiza mediante un lector fluorescente o una tira de flujo lateral. e, Lectura de ensayo de tira de flujo lateral. Un resultado positivo requiere la detección de al menos uno de los dos objetivos de genes virales de SARS-CoV-2 (gen N o gen E, como se indica en la matriz de interpretación). Control de calidad, control de calidad.
Luego probaron el ARN extraído de 11 muestras de torunda respiratoria recolectadas de seis pacientes con COVID-19 positivo para PCR (COVID19-1A / B a COVID19-5A / B, donde A se refiere a una torunda nasofaríngea y B se refiere a una torunda orofaríngea y COVID19- 6 se refiere a un solo hisopo nasofaríngeo) y 12 muestras de hisopos nasofaríngeos de pacientes con influenza (n = 5) e infecciones comunes por coronavirus estacionales en humanos (n = 7, que representan OC43, HKU1, 229E y NL63) utilizando el ensayo DETECTR SARS-CoV-2 con lecturas de tira de flujo lateral y a base de fluorescencia.
- El SARS-CoV-2 se detectó en 9 de 11 hisopos de pacientes y no reaccionó de forma cruzada con otros virus respiratorios. Se confirmó que dos hisopos negativos de pacientes con COVID-19 estaban por debajo del límite analítico de detección (LOD) establecido.
Dada la alta concordancia entre el flujo lateral y las lecturas basadas en fluorescencia (23 de 24 pruebas, o 95.8%), utilizaron una lectura basada en fluorescencia para analizar ciegamente 60 muestras adicionales de torunda nasofaríngea de pacientes con infección respiratoria aguda por SARS-CoV- 2 utilizando el ensayo DETECTR.
De las 60 muestras,
- 30 fueron positivas para la infección por COVID-19 mediante la prueba qRT-PCR
- y 30 fueron negativas para la infección por COVID-19 pero positivas para otra infección respiratoria viral por la prueba de PCR multiplex del panel de virus respiratorio o negativas para todas las pruebas.
Comparación del ensayo DETECTR (RT-LAMP / Cas12) con el ensayo CDC qRT-PCR para la detección de SARS-CoV-2
|
|
ARS-CoV-2 DETECTR, RT–LAMP/Cas12 |
CDC SARS-CoV-2 qRT–PCR |
|
Objetivo |
Gen E y gen N a |
Gen N (three amplicons, N1, N2 and N3) |
|
Muestra control |
RNase P |
RNase P |
|
Límite analítico de detección (LOD) |
10 copies per µl input |
1 copy per μl inputb and 3.2 copies per µl inputc |
|
Duración (aproximada) |
30–40 min |
120 min |
|
Duración desde la muestra al resultado (aproximada) |
45 min (with manual RNA extraction) |
4 horas (including RNA extraction) |
|
Resultado del análisi |
Cualitativa |
Cuantitativa |
|
Componentes del análisis |
RT–LAMP (62 °C, 20–30 min) |
UDG digestion (25 °C, 2 min), reverse transcription (50 °C, 15 min), denature (95 °C, 2 min) amplification, (95 °C, 3 s; 55 °C 30 s; 45 cycles) |
|
¿Se requiere instrumentación voluminosa? |
No |
Sí |
|
Aprobación de US FDA y EUA |
Pendiente de validación clínica |
Sí |
a Los cebadores del gen E se dirigen a la misma región de amplicón que en el protocolo de la OMS; Los cebadores de genes N se dirigen a la misma región de amplicón N2 que en el protocolo CDC. UDG, uracilo-ADN glicosilasa.
b Límite de confirmación de detección CDC 2019-nCoV Panel de diagnóstico de RT-PCR en tiempo real con QIAGEN QIAmp DSP Viral RNA Mini Kit6.
c Límite de confirmación de detección del panel de diagnóstico de RT-PCR en tiempo real CDC 2019-nCoV con QIAGEN EZ1 DSP6.
VENTAJAS
- Estudia dos genes: E y N
- Menor duración de la prueba
- No requiere equipamiento voluminoso
LIMITACIONES
- Todavía es una prueba experimental. Tomará aproximadamente dos semanas desarrollar la prueba CRISPR para uso en laboratorio clínico, y que una versión de punto de atención podría estar lista en tan solo dos o tres meses
- La técnica RT-PCR tiene la ventaja de procesar 386 muestras [a la vez], lo cual no es el caso con este método CRISPR.
- La prueba tampoco puede alcanzar el mismo nivel de sensibilidad que se ve en RT-PCR utilizada por los CDC, y aún queda trabajo por hacer para determinar su idoneidad para una implementación más grande y la aprobación de la FDA,
REFERENCIA
Broughton JP, Deng X, Yu G, et al. A CRISPR–Cas12-based assay for detection of SARS-CoV-2. Nat Biotechnol. 2020 Apr 16. doi: 10.1038/s41587-020-0513-4
PROF. DR. FERNANDO GALAN