Fernando Galán Galán
Profesor Titular de Medicina
Especialista en Medicina Interna
Experto en Miopatía Mitocondrial del Adulto
Fibromialgía y Síndrome de Fatiga crónica
BLOG
ENTENDIENDO LA NUEVA VARIANTE DEL COVID-19 DETECTADA EN EL REINO UNIDO.
La nueva variante, denominada linaje B.1.1.7, 20B/501Y.V1 o VOC–202012/01, incluye una combinación de 17 mutaciones diferentes que dan lugar a cambios en proteínas espiga (S) del virus. Algunas de estas mutaciones ya se habían encontrado anteriormente de forma individual, pero no en una misma versión del virus.
Propiedades genómicas de la nueva variante B.1.1.7, 20B/501Y.V1 o VOC–202012/01del SARS-CoV-2
- Esta nueva variante del virus SARS-CoV-2 se conoce en el Reino Unido como SARS-CoV-2 VUI 202012/01.
- Se define por múltiples mutaciones en la proteína espiga (deleción 69-70, deleción 144, N501Y, A570D, D614G, P681H, T716I, S982A, D1118H) presentes así como mutaciones en otras regiones genómicas. Una de las mutaciones (N501Y) se encuentra dentro del dominio de unión al receptor.
- La variante pertenece al clado Nextstrain 20B , clado GISAID GR, linaje B.1.1.7.
- Debido a la diversidad genética en expansión natural de los virus hCoV-19, GISAID introdujo un sistema de nomenclatura para los principales clados, desarrollado por Sebastian Maurer-Stroh et al, basado en mutaciones de marcadores dentro de 6 agrupaciones filogenéticas de alto nivel de la división temprana de S y L , a la posterior evolución de L en V y G y más tarde de G en GH y GR.
- Clado: se le llama clado a cada una de las ramas del árbol filogenético
Dentro de las mutaciones llama especialmente la atención que ocho de ellas se encuentran localizadas en la proteína S, proteína esencial para la entrada del virus a las células humanas. Dada la relevante función de la proteína S en el proceso de infección del coronavirus, la aparición de mutaciones en su secuencia tiene potencial para influir en la capacidad infectiva del virus.
PROPAGACIÓN
El linaje B.1.1.7 (también llamado 501Y.V1) es un grupo filogenético que se está extendiendo rápidamente en el sureste de Inglaterra. Había acumulado 17 mutaciones que definen el linaje antes de su detección a principios de septiembre, lo que sugiere una cantidad significativa de evolución previa, posiblemente en un huésped con infección crónica.
Al 28 de diciembre de 2020, esta variante representaba aproximadamente el 28% de los casos de infección por SARS-CoV-2 en Inglaterra, y los modelos genéticos poblacionales sugieren que se está propagando un 56% más rápidamente que otros linajes.
A diferencia de la mutación D614G, que plausiblemente podría haberse beneficiado de eventos al azar tempranos, el linaje B.1.1.7 se expandió cuando los casos de SARS-CoV-2 se generalizaron y aparentemente ha logrado el dominio al superar a una población existente de variantes circulantes. Esto sugiere fuertemente la selección natural de un virus que es más transmisible a nivel de población.
Si bien las intervenciones de salud pública como las máscaras, el distanciamiento físico y las limitaciones de las grandes reuniones deben seguir siendo efectivas, el control de esta variante más transmisible probablemente requeriría una aplicación más estricta y una adopción generalizada de estas medidas.
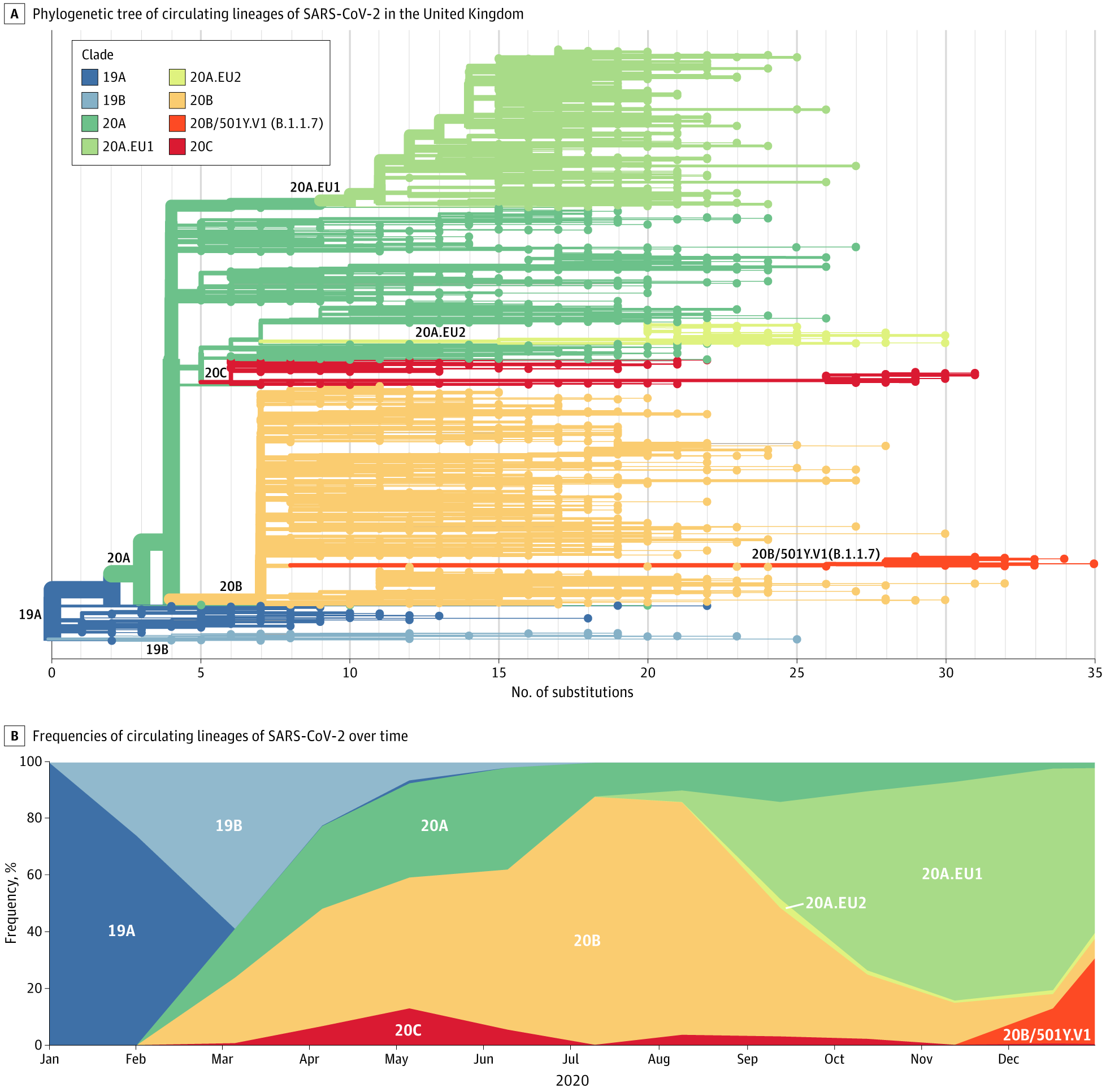
A, árbol filogenético que muestra la relación del linaje B.1.1.7 (20B / 501Y.V1, rama y puntas de naranja) con otros linajes circulantes. La larga longitud de la rama de este linaje refleja el hecho de que acumuló un número significativo de mutaciones antes de ser descubierto.
B, Frecuencias de los linajes circulantes a lo largo del tiempo. Los linajes están coloreados como en el árbol, con el linaje B.1.1.7 (20B / 501Y.V1) mostrado en naranja.
MUTACIÓN PROTEINA ESPIGA D614G
Técnicamente, D614G es una mutación del virus COVID-19 que causa el SARS-COV-2, presente entre la proteína espiga (S) del virus, que se utiliza para ingresar en las células del huésped. La mutación del ARN viral cambia el aminoácido D (que significa ácido aspártico) en "614" a G (glicina). Por tanto, la mutación está marcada como D614G (614 D>G). Haciendo más fácil la infección
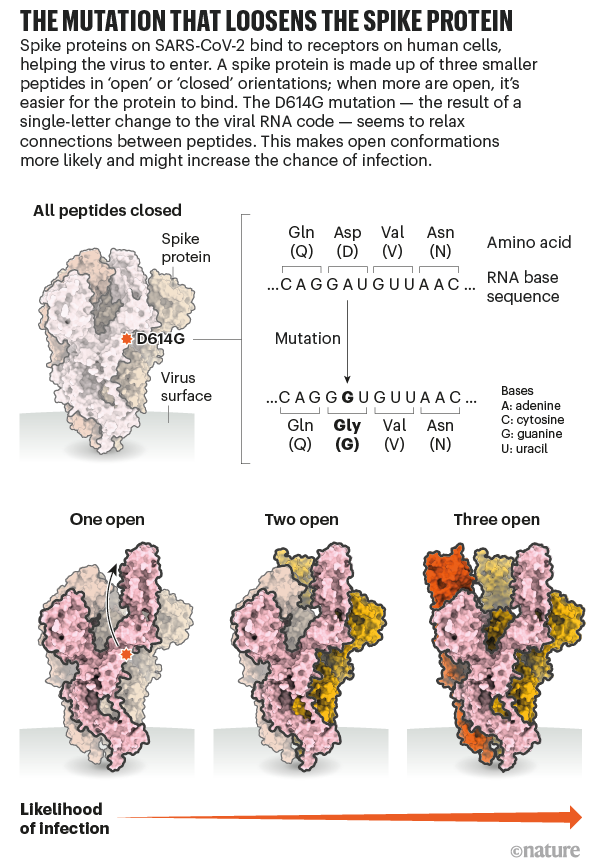
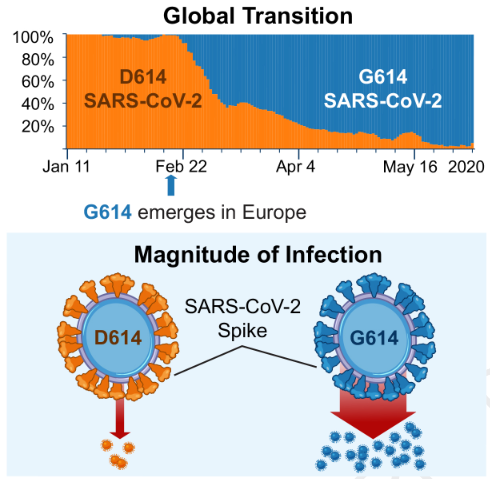
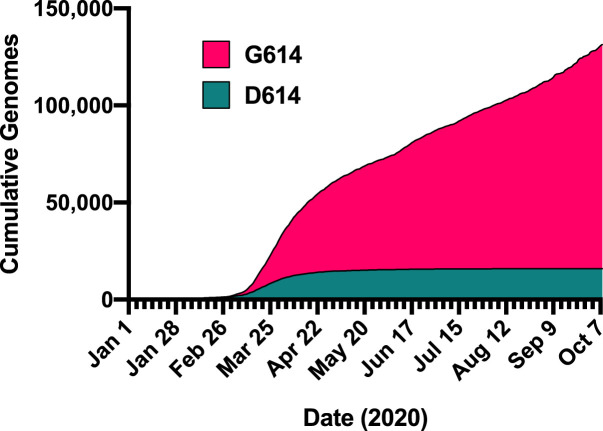
La mutación D614G en la glicoproteína de pico de SARS-CoV-2 se detectó por primera vez en febrero en algunos lugares de Europa y a un nivel significativo a principios de marzo de 2020 y se extendió a un dominio global durante el mes siguiente. La mutación inicialmente pareció surgir de forma independiente y simultáneamente en múltiples regiones geográficas. Esta aparente evolución convergente sugirió selección natural y un beneficio adaptativo de D614G. Sin embargo, los esfuerzos de secuenciación posteriores identificaron la mutación D614G en virus en varias provincias chinas a fines de enero. Esto planteó la posibilidad de que la dispersión global de esta mutación podría haber resultado de eventos fundadores casuales, en los que los virus que albergan 614G simplemente iniciaron la mayoría de los eventos de transmisión temprana en múltiples ubicaciones.
Durante la pandemia actual, hasta ahora solo ha habido una mutación en la proteína espiga S, D614G, que confiere una aparente ventaja de aptitud para el virus.
¿Cómo se originó la nueva variante?
Una de las hipótesis que plantean los expertos, es que el virus haya adquirido las mutaciones a través de su evolución en pacientes infectados de forma crónica. La larga exposición e incluso algunos de los tratamientos podría haber sometido al virus a una fuerte presión selectiva forzándolo a generar mutaciones y seleccionar las más eficientes. Diversos estudios ya han reportado una elevada tasa de acumulación de mutaciones en poco tiempo en pacientes infectados de forma crónica y aunque este tipo de infección no es muy frecuente, podría haber representado un caldo de cultivo para la nueva variante.
Actualmente no hay evidencia que sugiera que la variante tenga algún impacto en la gravedad de la enfermedad o la eficacia de la vacuna
La evaluación de una nueva variante del SARS-CoV-2 debe incluir la evaluación de las siguientes preguntas: ¿La variante alcanzó prominencia a través de la selección natural o eventos fortuitos? Si la evidencia sugiere selección natural, ¿qué mutación (es) se están seleccionando? ¿Cuál es el beneficio adaptativo de estas mutaciones? ¿Qué efecto tienen estas mutaciones en la transmisibilidad y diseminación, antigenicidad o virulencia?
CONCLUSIONES
- La nueva variante B.1.1.7, 20B/501Y.V1 o VOC–202012/01, incluye una combinación de 17 mutaciones diferentes que dan lugar a cambios en proteínas del virus. Algunas de ellas ya se habían encontrado anteriormente de forma individual, pero no en una misma versión del virus.
- La mutación de la proteína espiga D614G (614 D>G) confiere al COVID-19 mayor infectividad, pero no mayor virulencia.
- Actualmente no hay evidencia que sugiera que esta variante tenga algún impacto en la gravedad de la enfermedad o la eficacia de la vacuna
- El origen de la nueva variante puede ser debido a mutaciones a través de su evolución en pacientes infectados de forma crónica
REFERENCIAS
- Jackson CB et al. Functional importance of the D614G mutation in the SARS-CoV-2 spike protein. BIOCHEM BIOPH RES CO. Enero 2021
- Lauring AS, Hodcroft EB. Genetic Variants of SARS-CoV-2—What Do They Mean? JAMA. Published online January 06, 2021. doi:10.1001/jama.2020.27124
PROF. DR. FERNANDO GALAN
ANEXO
Clado: se le llama clado a cada una de las ramas del árbol filogenético.
Dentro de estos clados principales, monitoreamos los "clados emergentes" potenciales, que etiquetaremos por su clado padre y la mutación o mutaciones de nucleótidos que los definen (por ejemplo, 19A / 28688C). Sin embargo, cabe señalar que estas mutaciones solo son significativas en el sentido de que definen el clado. Una vez que un subclade alcanza los criterios (suaves) de frecuencia, propagación y distinción genética, se le cambiará el nombre a un clado principal (hipotéticamente 19A / 28688C a 20D).
|
Clado |
Países primarios |
Mutaciones |
Frecuencia máxima |
|
19A |
Asia: China/Thailand |
Root clade |
65-47% Globally in Jan |
|
19B |
Asia: China |
C8782T T28144C |
28-33% Globally in Jan |
|
20A |
N America/Europe/Asia: USA, Belgium, India |
C14408T A23403G |
41-46% Globally Apr-May |
|
20B |
Europe: UK, Belgium, Sweden |
G28881A G28882A G28883C |
19-20% Globally Mar-Apr |
|
20C |
N America: USA |
C1059T G25563T |
19-21% Globally Apr |
Actualmente, el clado G y su descendencia, GH y GR, son los clados más comunes entre los genomas secuenciados del SARS-CoV-2, y representan globalmente el 74% de todas las secuencias mundiales. Específicamente, el clado GR, que lleva la combinación de mutaciones de Spike D614G y Nucleocapsid RG203KR, es actualmente el representante más común de la población de SARS-CoV-2 en todo el mundo. La cepa viral original, representada por el clado L, todavía representa el 7% de los genomas secuenciados, y los otros clados derivados S y V tienen frecuencias similares en el conjunto de datos global.
|
Posición en el genoma del SARS-CoV-2 |
Intercambio de nucleótidos |
Frecuencia |
Región |
Intercambio de aminoácidos |
|
241 |
C→T |
70.99% |
5’ UTR |
-* |
|
1059 |
C→T |
18.10% |
Nsp2/ORF1ab |
T266I |
|
3037 |
C→T |
29.27% |
Nsp3/ORF1ab |
-* |
|
11083 |
G→T |
12.23% |
Nsp6/ORF1ab |
L36F |
|
14408 |
C→T |
70.42% |
Nsp12/ORF1ab |
P323L |
|
14805 |
C→T |
10.01% |
Nsp12/ORF1ab |
-* |
|
23403 |
A→G |
70.47% |
S Protein |
D614G |
|
25563 |
G→T |
22.49% |
ORF3a |
Q57H |
|
28881 |
G→A |
26.30% |
N Protein |
R204K |
|
28882 |
G→A |
26.13% |
N Protein |
|
|
28883 |
G→C |
26.11% |
N Protein |
G205R |
Las variaciones superiores al 20% de frecuencia se muestran en negrita *
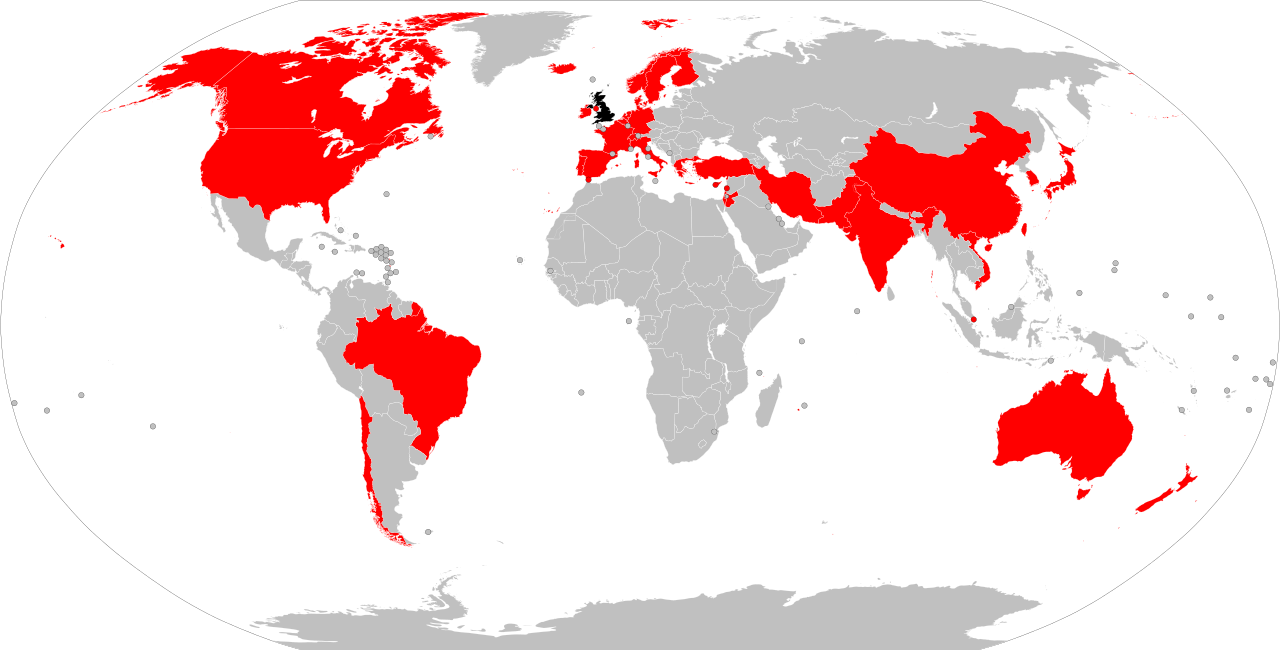
Mapa a 27 Diciembre 2020 de Países con casos confirmados de B.1.1.7, 20B / 501Y.V1 o VOC – 202012/01
Al 20 de diciembre de 2020, Public Health England confirmó que "no hay evidencia" que sugiera que la nueva variante sería resistente a la vacuna Pfizer-BioNTech que se está utilizando actualmente.




