Fernando Galán Galán
Profesor Titular de Medicina
Especialista en Medicina Interna
Experto en Miopatía Mitocondrial del Adulto
Fibromialgía y Síndrome de Fatiga crónica
BLOG
DESDE EL EDITOR GENETICO CRISPR - Cas9 AL NUEVO EDITOR DE LA BASE CITOSINA PARA CORREGIR GENES DEL ADN MITOCONDRIAL
El sistema de edición del genoma CRISPR-Cas9 ha permitido a los investigadores ajustar genomas a su gusto en casi todos los organismos en los que se ha probado. Pero la herramienta utiliza una cadena de ARN para guiar la enzima Cas9 a la región de ADN que los científicos desean editar. Esto funciona bien para el ADN en el núcleo de la célula, pero los investigadores no tienen forma de transportar ese ARN a las mitocondrias, que están rodeadas de membranas.
El ADN mitocondrial en una célula codifica solo 13 proteínas, todas las cuales están involucradas en la generación de energía de la célula. Las mutaciones en el ADN mitocondrial (ADNmt) causan una variedad de enfermedades mitocondriales. Por lo tanto, el desarrollo de herramientas para editar ADNmt ha sido un objetivo largamente buscado en la genética mitocondrial.
Recientemente ha sido publicado en Nature, Mok et al. una herramienta molecular que por primera vez permite la edición precisa de ADNmt. La clave de este logro fue el descubrimiento de una toxina secretada por bacterias para matar las bacterias vecinas. Surgió cuando Joseph Mougous, de la Universidad de Washington, había descubierto una enzima extraña. Era una toxina producida por la bacteria Burkholderia cenocepacia, que convertía la base C (citosina) del ADN en la base U (uracilo). Debido a que la base U, que no se encuentra comúnmente en el ADN, se comporta como una T (timina), las enzimas que replican el ADN de la célula lo copian como una T (timina), convirtiendo efectivamente una C en la secuencia del genoma en una T.
- Esta toxina puede matar a otras bacterias cambiando directamente la citosina (C) a uracilo (U) en el ADN bicatenario.
La toxina bacteriana descubierta por Mok et al. es una enzima citidina desaminasa llamada DddA, que cataliza la conversión de la base de nucleótidos citosina (C) a otra base, uracilo (U). Una característica notable de DddA es que se dirige al ADN bicatenario, mientras que todas las citidina desaminasas identificadas previamente se dirigen al ADN monocatenario.
Aunque los enfoques convencionales de edición del genoma involucran enzimas nucleasas que actúan como tijeras moleculares para cortar el ADN en ambas cadenas, DddA convierte C a U sin inducir roturas de ADN de doble cadena. Esto lo hace particularmente adecuado para editar el genoma mitocondrial, que carece de mecanismos eficientes para reparar roturas de ADN de doble cadena.
El ADN base Timina. ARN en la base Uracilo
Los investigadores tuvieron que superar varios desafíos para reutilizar DddA para la edición del genoma mitocondrial. El principal de ellos es el hecho de que la citidina desaminasa es tóxica para las células de mamíferos.
Mok y col. divide el dominio de toxina de DddA en dos partes inactivas llamadas mitades split-DddAtox. Fusionaron estas mitades con las proteínas TALE, que pueden diseñarse para unirse a secuencias de ADN específicas. La unión de los dos TALE al ADNmt reúne y, por lo tanto, activa las mitades DddAtox divididas.
Para alcanzar el ADNmt en la matriz mitocondrial, TALE-split-DddAtox debe cruzar dos membranas mitocondriales. Por lo tanto, Mok y sus colegas etiquetaron la construcción con una secuencia de aminoácidos que actúa como una señal de objetivo mitocondrial. La capacidad de explotar la maquinaria de importación de proteínas existente le da a este enfoque una gran ventaja sobre los sistemas guiados por ARN para la edición del genoma, como CRISPR-Cas9. Los métodos CRISPR no funcionan de manera eficiente en el ADNmt, posiblemente porque la célula no tiene mecanismos para importar ARN a las mitocondrias
El constructo final, denominado editor de base de citosina derivado de DddA (DdCBE), por lo tanto, consiste en una señal de objetivo mitocondrial, una proteína TALE, una mitad DddAtox dividida y un UGI. Mok y col. demostró que el constructo se importa eficientemente a las mitocondrias en las células humanas y puede modificar una selección de genes mitocondriales. La edición, de un par de bases C – G a T – A, se produjo entre el 5 y el 50% de las veces. La eficiencia de la edición estuvo influenciada por varios factores: el espacio entre las dos subunidades DdCBE; diseño TALE; orientación de las mitades split-DddAtox; y la posición de la citosina objetivo en relación con los sitios de enlaces TALE.
El equipo probó DdCBE en cinco genes en el genoma mitocondrial en células humanas y descubrió que DdCBE instaló ediciones de bases precisas en hasta el 50 por ciento del ADN mitocondrial. Se centraron en el gen ND4, que codifica una subunidad del complejo enzimático mitocondrial I, para una mayor caracterización. El laboratorio de Mootha analizó la fisiología mitocondrial y la química de las células editadas y mostró que los cambios afectaron a las mitocondrias según lo previsto.
Una herramienta para corregir el genoma mitocondrial.
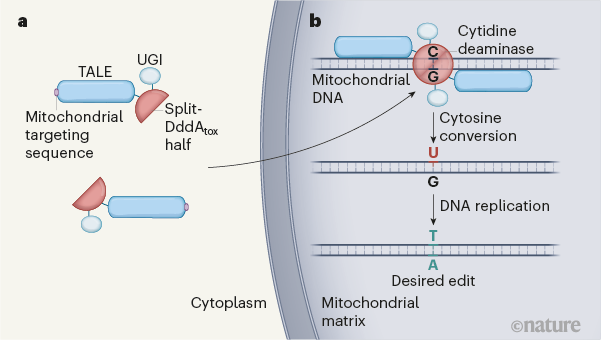
Mok et al. han desarrollado una herramienta molecular, DdCBE, que cambia los pares de bases de citosina y guanina (C – G) a pares de timina y adenina (T – A) en el ADN mitocondrial de la célula.
- Según el enfoque de los autores, DdCBE se divide inicialmente en dos construcciones, cada una de las cuales contiene: una porción inactiva de una toxina bacteriana, llamada división DddAtox; una proteína TALE que se une a una secuencia de ADN mitocondrial específica; una secuencia de aminoácidos que dirige la construcción a la matriz mitocondrial; y una proteína inhibidora de uracilo glucosilasa (UGI).
- La secuencia de direccionamiento mitocondrial se pierde cuando la construcción se importa a la matriz mitocondrial. Las proteínas TALE se unen a las secuencias de ADN mitocondrial adyacentes, uniendo y activando las dos mitades DddAtox divididas. La toxina actúa como una enzima que convierte la base C (citosina) objetivo en otra base, el uracilo (U). Normalmente, U se elimina del ADN, pero la proteína UGI protege la base editada hasta que se produce la replicación del ADN . En este punto, se reemplaza con T, que forma un par con A (adenina).
CONCLUSIONES
- Este nuevo editor de la base citosina proporciona un método para crear modelos de estudio de enfermedades causadas por mutaciones en el genoma mitocondrial. Hasta el momento, los modelos dependían de las mutaciones identificadas en pacientes o en la naturaleza y no se podían obtener nuevos.
- Respecto a su potencial en el desarrollo de tratamientos para enfermedades genéticas, todavía es muy pronto para plantear cualquier aplicación terapéutica.
REFERENCIA
Mok BY, et al. A bacterial cytidine deaminase toxin enables CRISPR-free mitochondrial base editing. Nature. 2020. Doi: https://doi.org/10.1038/s41586-020-2477-4
TRADUCCIÓN, RESUMEN Y COMENTARIOS:
PROF. DR. FERNANDO GALAN




